 世の中のことをもっと知るにはどうしたら良いだろうと思うときがある。世の中の多くの事柄はログやデータに落とされる。Googleなどの検索サイトは良い例だろう。さて、そのログやデータをどうすれば良いのか? 多くの場合、視覚化が有効な手段となる。
世の中のことをもっと知るにはどうしたら良いだろうと思うときがある。世の中の多くの事柄はログやデータに落とされる。Googleなどの検索サイトは良い例だろう。さて、そのログやデータをどうすれば良いのか? 多くの場合、視覚化が有効な手段となる。まずは身の回りの日常的なデータやログを何とかしたい。ただ、日常のデータを視覚化するのに数十行以上のコードは書きたくない。まるで息をするかのごとく自然に視覚化を行いたいのだ。そのためには1~2行、長くて数行で済ませることが必要だ。そこでPythonとmatplotlibを使う。加えて、IPythonがあればなお良い。IPythonの導入については以前のブログ記事であるIPythonの埋め込みプロットが素晴らしいを参考にして欲しい。
まずは事前にnumpyとmatplotlibをインポートしておく。できればscipyも。
>>> from numpy import * >>> from pylab import *
短いコードで視覚化を行うためには、Pythonの内包表記は必須だ。例えば、5, 2, 1, 5, 8をデータとするグラフを書きたいのならIPythonを使って以下の1行で実現できる。
>>> plot([5, 2, 1, 5, 8])
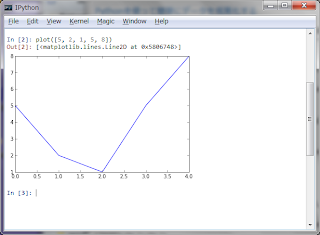
数値が行ごとにinput.txtというファイルに書かれていた場合は以下の通り。
>>> plot([int(x) for x in file("input.txt")])
map関数を使えばもっとスマートに書ける。ファイル内の数値を文字列として配列で読み込み、それらの文字列をmap関数によりintで整数に変換する。
>>> plot(map(int, file("input.txt")))
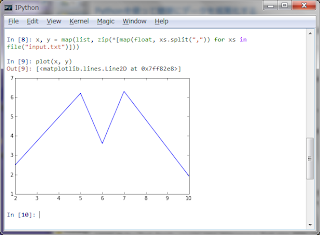
さて、ログが2次元で書かれていた場合はどうするのか。例えば以下のデータがinput.txtに書かれていたとする。
2, 2.5 5, 6.2 6, 3.6 7, 6.3 10, 1.9
この場合、以下のようにすればプロットできる。ファイルから行ごとの文字列を読み出して、それを","を区切りとしてリスト化する。文字列で格納されている数値をmap関数を使ってfloatで実数に変換している。最後にmap(list, zip(*data))を使って転置している。ここでdataはリストによる行列とする。リストによる行列の転置はイディオムとして覚えておくと便利だ。
>>> x, y = map(list, zip(*[map(float, xs.split(",")) for xs in file("input.txt")])) >>> plot(x, y)
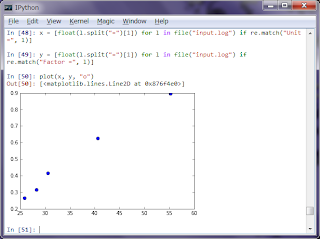
ログファイルなどの場合、単に数値が並んでいることは少なく一緒にテキストが書き込まれていることが多い。例えば次のログ(input.log)について視覚化を行なってみる。
Data analysis log: Cycle 1: Calculating ... done! Factor = 0.265 Unit = 25.8 Cycle 2: Calculating ... done! Factor = 0.315 Unit = 28.2 Cycle 3: Calculating ... done! Factor = 0.415 Unit = 30.5 Cycle 4: Calculating ... done! Factor = 0.625 Unit = 40.6 Cycle 5: Calculating ... done! Factor = 0.895 Unit = 55.2
Unitの値をそのままプロットするなら以下のようにすれば良い。予めreモジュールをインポートしておき、re.matchで先頭の文字列と一致したものだけ選び出している。
>>> plot([float(l.split("=")[1]) for l in file("input.log") if re.match("Unit =", l)])

Unitを横軸、Factorを縦軸にしてドットでプロットしたい場合は以下のようにする。
>>> x = [float(l.split("=")[1]) for l in file("input.log") if re.match("Unit =", l)] >>> y = [float(l.split("=")[1]) for l in file("input.log") if re.match("Factor =", l)] >>> plot(x, y, "o")
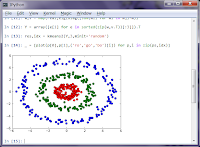
最後に少し複雑な例を挙げておく。あるデータに対してスペクトラルクラスタリングを行いたいとする。まずは、numpyとmatplotlibの他に、scipy関連の固有値問題・k近傍法・kd木のモジュールをインポートしておく。
>>> from scipy.linalg import eig >>> from scipy.cluster.vq import kmeans2 >>> from scipy.spatial.kdtree import KDTree
そして、クラスタリングしたいデータをcircles.datとすると、以下のコードで視覚化できる。
>>> ps = array([map(float,l.split()) for l in file("circles.dat")]) >>> kt = KDTree(ps) >>> knn = [[((lambda a,b:exp((-(sqrt(dot(a-b,(a-b).conj()))**2))/1.5))(p,ps[nb]),nb) for nb in kt.query(p,16)[1] if i!=nb] for i,p in enumerate(ps)] >>> W = zeros([len(knn)]*2) >>> _ = [W[p].__setitem__(nb,d) for p,nn in enumerate(knn) for d,nb in nn if p in zip(*knn[nb])[1]] >>> w,v = map(real,eig(diag([sum(Wi) for Wi in W])-W)) >>> Y = array([e[1] for e in sorted(zip(w,v.T))[:3]]).T >>> res,idx = kmeans2(Y,3,minit='random') >>> _ = [plot(p[0],p[1],('ro','go','bo')[i]) for p,i in zip(ps,idx)]
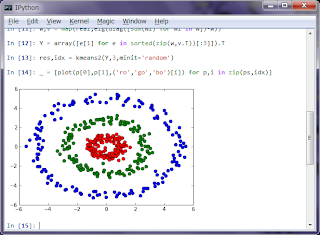
ここではforループを行うために内包表記を使っており、また、内包表記のリストが必要ない場合は、_(アンダースコア)に入れておく。これはシェル上で余計なデータを表示させないためである。また、内包表記上では配列への代入ができないので__setitem__を利用している。さらに、Pythonのリストの代わりにnumpyのarrayを利用すれば転置などもmap(list, zip(*data))の代わりにarray(data).Tとすれば良いので分かりやすい。
Pythonを使えばたったこれだけだ。これだけのことで、これまで見えなかったものが見えてくるのは楽しい。
コメント